昆明植物所在植物基因组LTR类转座子分类方面取得新进展
转座子是构成基因组重复序列的主要成分。越来越多的研究表明,转座子在决定基因组大小、基因组结构变异、序列突变、基因丢失、基因融合和新编码基因的起源方面,都具有重要的生物学意义。LTR类转座子是植物基因组中占比最高的重复序列类型,它是逆转录转座子的一种。然而目前大部分软件对LTR类转座子仅停留在超科水平(superfamily level),没有提供更细致的分类,无法反映LTR类转座子的多样性和进化关系。
基于此,研究团队开发出LTR类转座子更加准确、快速分类的新方法,命名为TEsorter。该方法是以已经分类的保守蛋白结构域(数据库来源REXdb或GyDB)为数据基础,采用hidden Markov models (HMMs)方法,对玉米和水稻基因组中转座子,尤其是LTR类转座子进行了准确分类。且大部分LTR类转座子可达到分支水平(clade level),分支水平的分类结果和基于系统发育树的聚类高度一致。同时,通过对比现在常用的5个转座子分类软件(RepeatModeler, DeepTE, TERL, LTR_retriever 和 LTRclassifier),TEsorter不论在准确率还是运算速度方面,都具有明显优势。研究成果以TEsorter: an accurate and fast method to classify LTR-retrotransposons in plant genomes为题在线发表于园艺学Top 1期刊、中科院1区期刊Horticulture Research上。
本研究在云南省基础研究专项重大项目(202101BC070003)、云南省中青年学术与技术带头人(2018HB066)、支持下,由中国科学院昆明植物研究所极小种群野生植物综合保护研究大团队马永鹏专题组、源宜基因张仁纲研究团队和爱荷华州立大学区树俊研究团队合作完成。
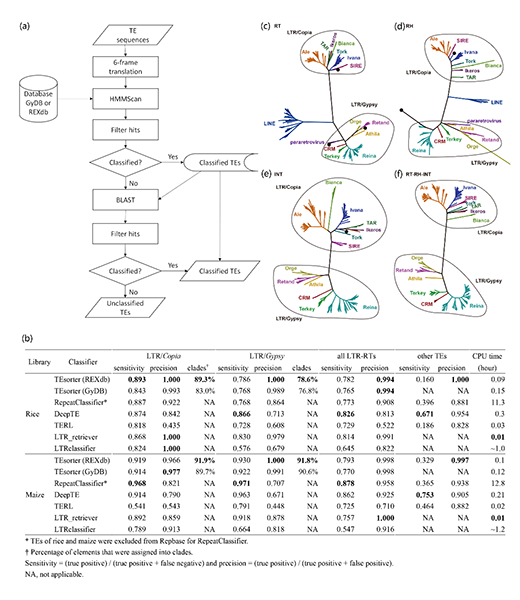
图 (a) TEsorter计算流程; (b) TEsorter与目前5个常用转座子分类软件(RepeatModeler, DeepTE, TERL, LTR_retriever 和 LTRclassifier)在敏感度、准确率及计算时间等参数方面的比较;(c-f ) TEsorter分类结果和基于系统发育方法对水稻和玉米中的LTR类转座子分类的一致性比较。
